纤维小体是细菌分泌的高效降解木质纤维素的多酶复合体,其高效降解机制及产纤维小体细菌的遗传改造是木质纤维素降解利用研究中的重要方向之一。热纤梭菌的Cel48S是其纤维小体的主要外切葡聚糖酶,是其纤维小体中含量最高的组分,在纤维素降解过程中起着关键作用。但是,Cel48S的内在性质使得对Cel48S的纯化存在很大的困难,文献中对该酶的性质鉴定报道存在很多相互矛盾之处,与该酶在纤维小体中的重要作用并不相符,这使人们难以深入揭示纤维小体的高效降解机制。青岛能源所代谢物组学团队崔球研究员、冯银刚研究员、刘亚君副研究员等基于前期开发的热纤梭菌遗传改造工具,通过对纤维小体的定向改造,实现了对其原位Cel48S催化结构域的高效纯化,从而成功的对Cel48S的酶学与结构性质进行了系统解析,揭示了Cel48S在纤维小体中的真正作用。相关成果已于2018年1月13日在线发表于Biotechnology for Biofuels (Liu YJ, et al, 2018, 11:6)。
木质纤维素水解糖化是实现木质纤维素生物质的高值利用的第一步,是国内外的研究热点。为了打破国外酶制剂技术垄断、突破糖化技术瓶颈,代谢物组学团队长期致力于开发基于热纤梭菌纤维小体的新型整合生物糖化技术路线。在这一路线框架下,需要通过对纤维小体这一多酶复合体的机制解析以及定向改良从而获得高效的全菌催化剂。为此,代谢物组学团队针对梭菌系统地开发了非模式微生物基因操作工具(J Microbiol Methods, 2012, 89: 201-8.; PloS One, 2013, 8:e69032; Appl Microbiol Biotechnol, 2014, 98: 313-23.),在热纤梭菌中实现了快速、准确的基因敲除、过表达和精准编辑,完成了对热线梭菌纤维小体脚架蛋白的功能贡献、解除纤维小体反馈抑制的定向改造(Biotechnol Biofuels, 2015, 8: 36; Biotechnol Biofuels 2017, 10: 124)等系列工作。同时,团队建立了成熟的X射线晶体学和生物大分子核磁共振等蛋白质结构解析平台,完成了多个纤维小体组分和工业酶催化机制的研究(J Struct Biol, 2014, 188: 188-193; Appl Microbiol Biotechnol, 2015, 100: 2203–2212)。在完善的遗传操作平台和蛋白质结构解析平台的基础之上,研究人员对纤维小体关键组分Cel48S进行了更系统深入的研究。
作为热纤梭菌纤维小体中含量最高的的关键酶组分,Cel48S的酶学性质及结构特点一直受到广泛的关注。团队前期通过核磁共振方法修正了文献中关于Cel48S的组装模块的结构,对纤维小体的组装方式有了新的认识(J Struct Biol, 2014, 188: 188-193)。对于Cel48S的催化模块,由于难于异源表达,与纤维小体结合紧密难以从原位分离,文献中对其性质的报道相互矛盾,该酶在纤维小体中的真正作用并未得到有效阐明。为了解决这一问题,研究人员使用前期建立的热纤梭菌精准遗传操作工具,通过在Cel48S的催化结构域后面插入编码组氨酸标签和终止密码子的序列,实现了将Cel48S的催化结构域从纤维小体中分离并从培养物上清中的一步直接纯化。在此基础上,通过酶学测定和蛋白质结构分析,确定了天然Cel48S的高活性、结晶纤维素的底物偏好以及底物偶联的诱导契合效应,真正彻底阐明了纤维小体关键组分Cel48S的结构功能特性。同时,本研究提供了一个基于精准遗传操作实现分离纯化原位的纤维小体或其他分泌蛋白的方法,为进一步研究纤维小体木质纤维素降解机制及构建木质纤维素糖化的定向改良菌株提供了范例和基础。
相关系列研究得到了国家科技支撑计划、国家自然科学基金委员会以及山东省糖产业科学技术重点实验室联盟建设任务的资助。(文/图 刘亚君 冯银刚)
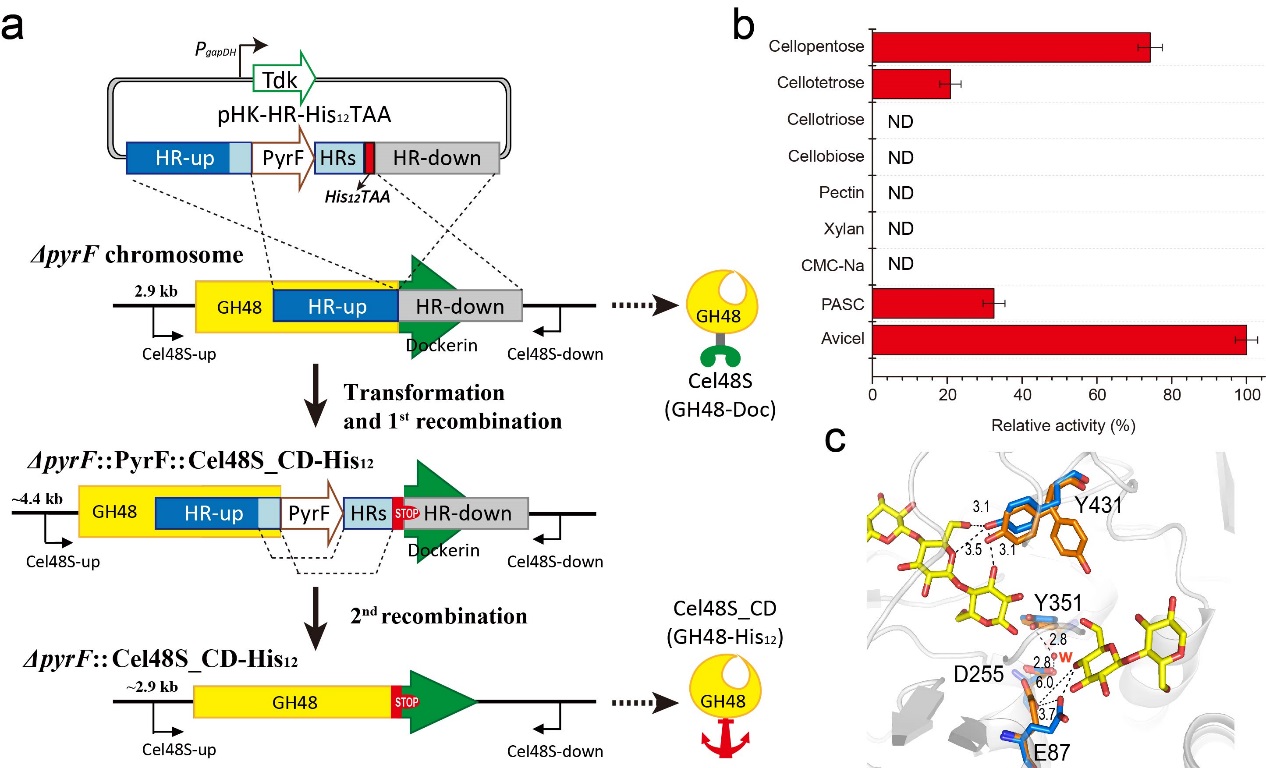
图1. 热纤梭菌Cel48S定向改造示意图(a)、Cel48S底物偏好性(b)及结构分析(c)
原文链接:
https://biotechnologyforbiofuels.biomedcentral.com/articles/10.1186/s13068-017-1009-4
Liu, Y.-J., S. Liu, S. Dong, R. Li, Y. Feng and Q. Cui (2018). "Determination of the native features of the exoglucanase Cel48S from Clostridium thermocellum." Biotechnology for Biofuels 11(1).