近日,实验海洋生物学重点实验室张琳琳团队在牡蛎基因组编辑方面获新进展,相关成果在学术期刊Frontiers in Marine Science发表。
随着对高品质动物蛋白需求的迅速增长,水产养殖正成为人类食用海产品的主要来源。然而,与许多成熟的陆生牲畜和作物系统相比,大多数水产养殖物种育种仍处于驯化的早期阶段。传统的育种方式,如选择、杂交和标记辅助育种系统,已经推进了水产养殖物种经济性状(包括抗病性、营养价值和生长质量等)的遗传改良。基因组编辑技术是近年来研究基因功能和解析性状最直接有效的方法,其中CRISPR/Cas9基因编辑技术因具有操作简单、靶点选择广、成本低、效率高等优点,为重要水产物种经济性状的遗传解析和良种培育提供了最直接有力的工具。
牡蛎为世界大宗水产养殖贝类,但和水产鱼类相比,基因组编辑育种技术在牡蛎中应用还处于起步阶段。针对牡蛎卵径小(~50 m)、显微操作难、幼虫死亡率高、间接发育时间时间长以及在获得可遗传纯系方面难度大、成本高、耗时长的难题,中科院海洋所张琳琳研究团队经过长期的研究攻关,搭建了一套基于电穿孔的Cas9/sgRNA复合物高通量递送和突变体快速检测的技术平台,成功实现了对牡蛎(Crassostrea gigas angulate)基因组中标记基因( -tubulin)的高效编辑。
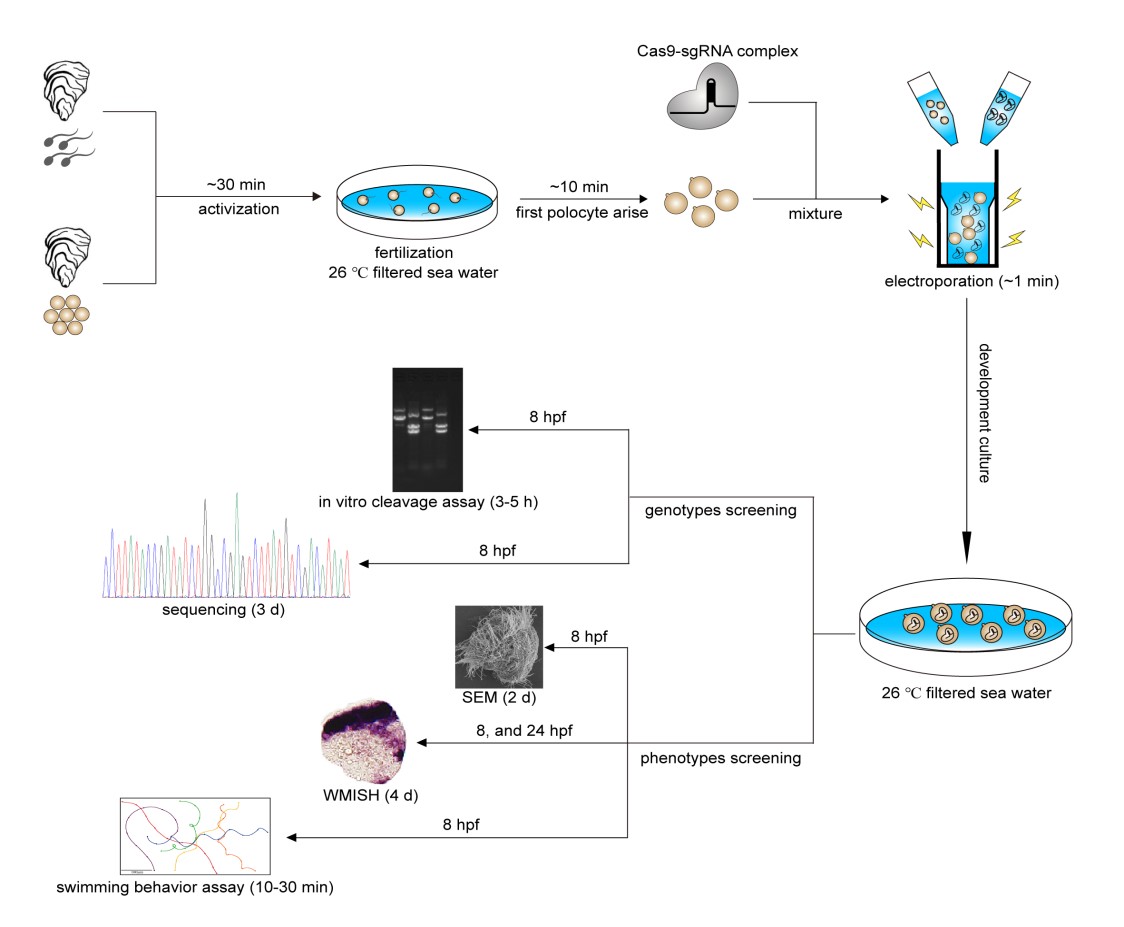
基于电穿孔的CRISPR/Cas9基因编辑系统和突变表型快速检测平台示意图
研究采用作者前期研发的“长片段缺失镶嵌性突变技术”(Zhang L., et al, 2016, Nature Communications; Zhang L., et al., 2017, PNAS)。通过同时电穿孔多个靶基因sgRNAs,研究人员在长牡蛎靶向基因中检测到超过300 bp的长片段缺失突变,这使得研究人员可以使用PCR和常规琼脂糖凝胶电泳对突变体进行快速筛选和基因分型。这种同时传递两个以上sgRNAs以获得长片段缺失的策略是一个显著的改进,大大简化了基因组编辑基因型检测的工作流程。此外,利用原位杂交和行为学分析等表型检测手段,研究人员在牡蛎G0代幼虫中观察到了表型的镶嵌型突变(纤毛的缩短、缺失)和运动能力下降。这种镶嵌型突变有利于研究人员在G0代个体中对突变表型进行快速的鉴别,同时也能规避目标基因完全缺失导致的胚胎致死性。该研究在海洋经济贝类牡蛎中建立了基于电穿孔和长片段缺失镶嵌性突变的CRISPR/Cas9基因编辑平台,可为今后基于CRISPR/Cas9基因组编辑技术在海洋贝类中开展基因功能研究提供有益的参考,同时也为牡蛎以及其他水产养殖物种的基因组编辑育种提供有力的工具。
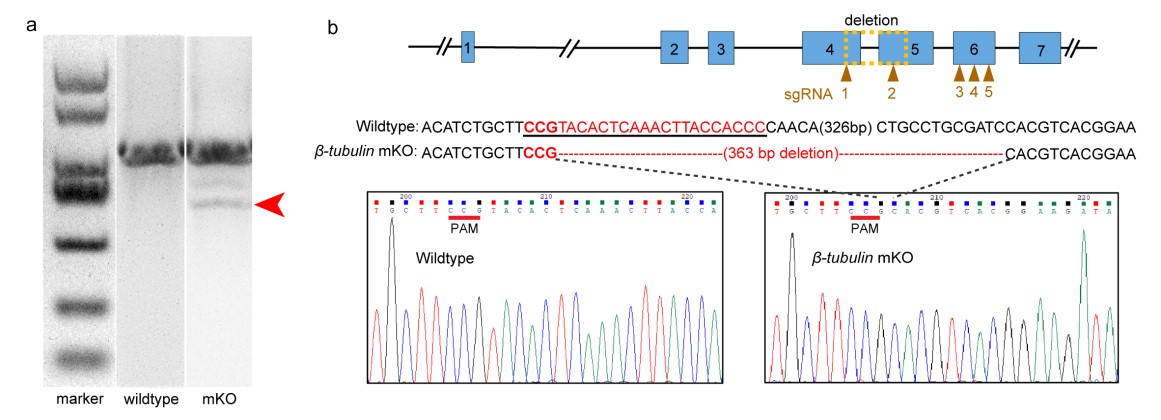
CRISPR/Cas9系统介导的长牡蛎 -tubulin基因的长片段缺失
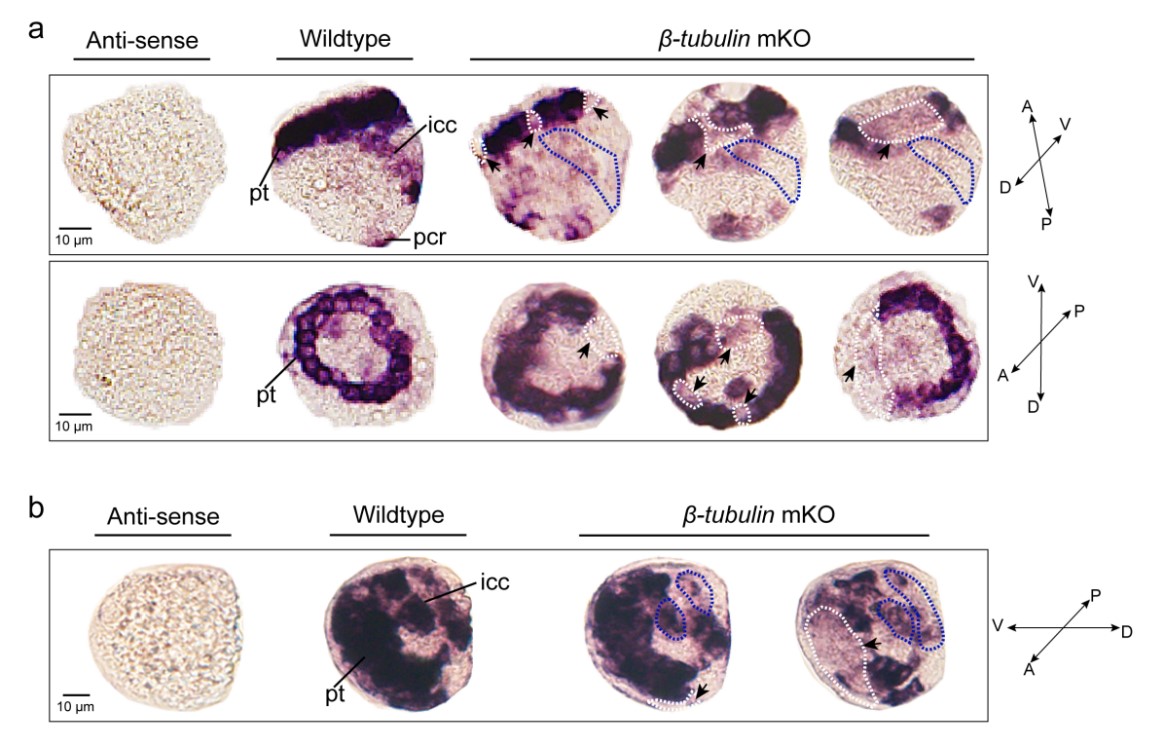
CRISPR/Cas9系统介导的长牡蛎镶嵌型突变的纤毛表型
海洋研究所实验海洋生物学重点实验室博士后产久林和硕士研究生张韦为论文的共同第一作者,张琳琳研究员为通讯作者,实验海洋生物学重点实验室科研助理许悦、研究生薛雨、吴富村副研究员、张国范研究员和李莉研究员参与了该项目。研究得到了山东省“海洋生命资源绿色发展技术与应用”工作站,中科院先导专项B和国家海外引才计划青年项目等项目的资助。
相关成果如下:
1.Chan, J.#, Zhang, W.#, Xu, Y., Xue, Y. & Zhang, L.* (2022). Electroporation-based CRISPR/Cas9 mosaic mutagenesis of -tubulin in the cultured oyster. Frontiers in Marine Science, 9: 912409. doi: 10.3389/fmars.2022.912409.
2.张琳琳,许悦,张韦,产久林。快速获得基因型和表型突变的CRISPR/Cas9基因敲除方法及应用,专利申请号202210378237.8。
3.张琳琳,张韦,许悦,产久林。长牡蛎 -tubulin基因的电穿孔基因编辑方法及应用,专利申请号202210378335.1。
4.张琳琳,许悦,吴富村。一种皱纹盘鲍CRISPR/Cas9基因编辑的方法,专利申请号202111053880.5。
5.Zhang, L., Mazo-Vargas, A. & Reed R.* (2017). A single master regulatory gene coordinates the evolution and development of butterfly color and iridescence. Proceedings of the National Academy of the USA, 114(40):10707-12.
6.Zhang, L. & Reed R.D.* (2016). Genome editing in butterflies reveals that spalt promotes and Distal-less represses eyespot colour patterns. Nature Communications, 7, 11769.